Les contaminations entre lignées cellulaires : un mal bien souvent ignoré…
Les cas de contaminations croisées entre lignées cellulaires sont beaucoup plus fréquents qu'on ne le pense. Une enquête menée dans la littérature scientifique a démontré qu'environ 20% des travaux publiés seraient basés sur l'utilisation de lignées cellulaires humaines mal identifiées (réf 1,2) .
Cet article a pour objectif d'attirer l'attention sur les lignées cellulaires connues pour être mal identifiées ou que l'on soupçonne de l'être. Pour voir la liste complète des lignées cellulaires mal identifiées, compilée par le Dr R. Freshney Ian et le Dr Amanda Capes-Davis cliquez ici . Cette liste a été réalisée sur la base de rapports publiés dans la littérature scientifique et les détails sont donnés par les banques cellulaires et leurs sites Web. Nous vous invitons également à lire la lettre ouverte du Dr Nardone, qui conseille à toute personne et organisations utilisant des lignées cellulaires de prendre des mesures appropriées.
Les informations ci-dessous sont spécifiques aux lignées cellulaires affectées par une mauvaise identification ou par une contamination croisée auparavant disponibles et maintenant retirées du catalogue ECACC.
Pour les lignées cellulaires à l'origine mal identifiées encore disponibles au catalogue, l'ECACC a effectué des tests permettant d'identifier l'origine réelle de ces lignées, et met en avant leur nouvelle identité en précisant qu'elle annule et remplace celle présumée initialement.
|
• Un archivage partagé des documents au sein du même laboratoire • Développer une démarche environnementale en réduisant l'utilisation de papier |
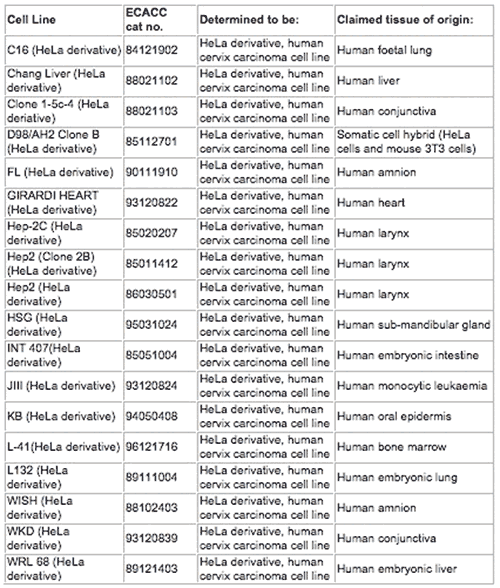
1.Les Dérivés de la lignée HeLa
Les lignées cellulaires énumérées dans le tableau 1 ci-dessous ont été montrées comme étant génétiquement indiscernables de la lignée cellulaire de carcinome du col de l'utérus humain HeLa lors de l'analyse des séquences « short tandem repeat ». Par conséquent, ces lignées cellulaires doivent être considérées comme des cellules HeLa. De ce fait, l'indication « Hela derivative » apparait à côté du nom d'origine de la lignée cellulaire lorsque ce dernier apparait dans le catalogue ECACC. De telles lignées cellulaires devraient être considérées comme des dérivés de tissus cervicaux et ne pas être utilisées dans des études pour représenter tout autre type de tissus, y compris pour reproduire des études attribuées à tort à la lignée cellulaire mal identifiée.
Tableau 1 :Lignées cellulaires connues pour être des dérivés de la lignée HeLa de carcinome du col de l'utérus humain. La contamination croisée s'est produite avant le dépôt auprès de l'ECACC et affecte toutes les sources connues de ces lignées cellulaires.
|
 |
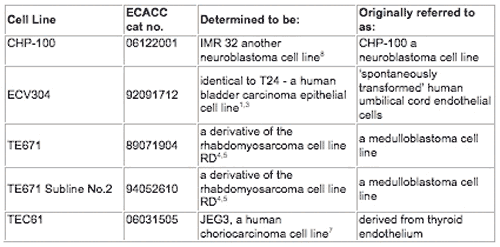
2. Les lignées cellulaires avec une nouvelle identité (non dérivées d'HeLa)
Tableau 2 : Les lignées cellulaires ci-dessus listées se trouvant être cross-contaminées ou identifiées à tort comme d'autres lignées cellulaires, ont vu leur identité réaffectée. Veuillez noter que ces nouvelles identités affectent tous les sources officielles des lignées cellulaires disponibles. La contamination croisée s'est produite avant le dépôt auprès de l'ECACC.
|
 |
3. En quête d'identité…
Lignée U-373 MG
L'authenticité de certaines sources de la lignée cellulaire U-373 MG a été remise en question6. L'ATCC a déclaré que leur lignée U-373 MG n'a pas les mêmes caractéristiques génétiques que la lignée décrite par le laboratoire d'origine, mais partage des similitudes avec la lignée cellulaire de glioblastome U-251. À la lumière de ces informations, l'ECACC a suspendu la distribution de la lignée cellulaire U-373 MG et a entrepris une enquête sur l'authenticité de son propre stock. L'ECACC a obtenu des résultats similaires à ceux de l'ATCC, c'est à dire que le stock considéré comme étant des U-373 MG a été jugé identique au niveau des séquences « short tandem repeat » à la lignée U-251. La lignée cellulaire U-373 MG, enregistrée sous la référence 8908140, a donc été renommée «U-251, antérieurement connu sous le nom U-373 MG » et a reçu une nouvelle référence au catalogue ECACC: 09063001. Un nouveau stock de U-373 MG a été obtenu auprès de l'auteur de la lignée d'origine : il montre bien une différence avec la lignée U-251 au niveau des séquences STR. La lignée a été ajoutée à la collection et est appelée U-373 MG (Uppsala), référence ECACC 08061901, pour la distinguer de la lignée cellulaire U-373 MG, référencée sous le numéro 89081403 et appelée «U-251 antérieurement connue sous le nom U-373 MG ». Les deux lignées cellulaires sont dérivées de glioblastomes astrocytaires.
La lignée IPTP/98
L'ECACC a récemment déterminé, en utilisant l'analyse d'espèce par le profil d'isoenzymes, que l'échantillon de cellules IPTP/98 déposé auprès de l'ECACC est originaire du rat et non de l'homme (Origine initiale contestée : lignée cellulaire humaine de glioblastome). En conséquence de cette découverte, la lignée IPTP/98 a été retirée du catalogue ECACC.
La lignée IPDDC-A2
L'ECACC a récemment déterminé, en utilisant l'analyse d'espèce par le profil d'isoenzymes, que l'échantillon de cellules IPDDC-A2 déposé auprès de l'ECACC est originaire du rat et non de l'homme (Origine initiale contestée: lignée cellulaire humaine d'astrocytome). En conséquence de cette découverte, la lignée IPDDC-A2 a été retirée du catalogue ECACC.
La lignée TEC61
L'ECACC a récemment déterminé à l'aide de l'analyse par PCR des séquences STR que la lignée TEC61 (référence ECACC 06031505), prétendue dérivé de l'endothélium de la thyroïde, est en réalité la lignée JEG3, un choriocarcinome humain. Le milieu de culture conditionné de la lignée JEG3 a été utilisé pour aider la croissance de la lignée cellulaire endothéliale putative (Patel et al, 2009 PMID: 12388152, rétraction - PMID:. 19625678) 7. Conséquence de cette découverte, la lignée TEC61 a été retirée du catalogue ECACC.
CHP-100
L'ECACC a récemment déterminé, à l'aide de l'analyse par PCR des séquences STR, que la lignée CHP-100 (référence ECACC 0612200), supposée être un neuroblastome, est en réalité la lignée IMR 32, un autre neuroblastome8. En conséquence de cette découverte, la lignée CHP-100 a été retirée du catalogue ECACC.
References
1. MacLeod et al., (1999) Widespread intraspecies cross-contamination of human tumor cell lines arising at source. Int J Cancer 83(4) 555-563 (Cell line: ECV304)
2. Drexler HG et al. (2003) False leukemia-lymphoma cell lines: an update on over 500 cell lines. Leukemia, 17(2):416-426.
3. Dirks et al., (1999) ECV304 (endothelial) is really T24 (bladder carcinoma): cell line cross- contamination at source In Vitro Cell Dev Biol Anim 35: 558-559 (Cell line: ECV304)
4. Stratton et al., (1989) Misidentified Cell. Nature;337:311; (Cell line: TE671)
5. Chen et al., (1989) Cell Identity Resolved. Nature;340:106; (Cell line: TE671)
6. ATCC website & Ishii et al., (1999) Frequent Co-Alterations of TP53, p16/CDKN2A, p14ARF, PTEN Tumor Suppressor Genes in Human Glioma Cell Lines. Brain Pathol 9: 469-79, (Cell line: U-373 MG)
7. Patel et al., 2003 Isolation and characterization of human thyroid endothelial cells. Am J Physiol Endocrinol Metab. 2003 Jan;284(1):E168-76. PMID: 12388152 Retraction (2009): PMID:19625678 (Cell line: TEC61)
8. Reported by the European Collection of Cell Cultures (ECACC) on the Health Protection Agency Culture Collections website. (Cell line: CHP-100)
Pour recevoir le Manuel de Culture cellulaire Sigma, cliquez ici
| Demande d'information |
